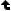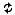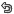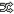cjftn0/2008-03-30
|
Describe cjftn0/2008-03-30
-미팅시간에 10cv의 평균이 70%정도라고 말씀드렸는데, 이번의 결과는 약간 낮게 나왔습니다. 이런 이유는 랜덤 샘플링에 의해서 초기의 샘플들을 n1,n2,.....,n10까지의 데이터 셋으로 나누게 되는데, 이 데이터 셋의 각 모수(mean, variance)가 매번 모델 만들 때 마다 변경되게 되고, 이러한 영향이 매번 결과에 영향을 주기 때문으로 생각됩니다.
앞으로 여러 번 모델을 만들어서 모델 정확도의 평균을 산출하여 결과에 포함시키겠습니다.
교수님 가르침으로 지금까지 간과 하고 있었던 부분을 새삼 깨닫게 되었습니다. ^^;;
-분류 모델 구성에 사용된 방식
1.msc.features.select 로 속성차원 줄임
2.tree 모델 - 사용된 feature selection 방식은 gini
1.속성 선택하는 다른 메서드 이용- relifcat feature selection
0.98/0.72 => null vector (선택된 속성이 없는 cv 발생)
0.98/0.75 => null vector (선택된 속성이 없는 cv 발생)
0.98/0.8 => null vector (선택된 속성이 없는 cv 발생)
결과- 10cv의 평균 약 58%
2.rpart 이용하여 모델 구축시도 -벡터할당 에러 or too many element specified
3.msc.features.select에서 RemCorrcol, keepCol 파라미터 조정
-시도 도중 얻은 아이디어
1.오분류율이 15% 이하인 10cv들 중에서 공통된 속성을 발견
이 속성이 사용 안 된 10cv는 오분류율이 높다는 것을 발견
빈발하는 속성의 빈도수 및 공통속성(5번의 빈도수 가진 속성)
=>오분류율이 15% 이하인 10cv들 중에서 선택된 속성들의 전체 집합에서 빈발하 는 속성과 공통속성을 이용하여 모델 구축.다음주 계획 5번의 10 cross validation을 통해 만들어진 10cv 중에서 약15% 이하의 오분류율을 나타 낸 10cv 만 선택하여, 이것의 선택 되어진 속성의 공통된 속성과 빈도수를 구했습니다.
속성
빈도수
"X228769_at"
공통속성
“X227094_at”
4
"X219821_s_at"
3
"X1557483_at"
3
"X1564190_x_at"
2
"X221572_s_at"
2
"X227356_at"
2
"X219429_at"
2
Classification tree:
tree(formula = class ~ ., data = iter10, na.action = na.pass,
split = c("gini"), x = FALSE, y = TRUE)
Variables actually used in tree construction:
1 "X219821_s_at" "X1557483_at" "X227094_at" "X212707_s_at"
5 "X234668_at" "X220455_at" "X206819_at" "X1564190_x_at"
9 "X228769_at"
Number of terminal nodes: 13
Residual mean deviance: 0.5868 = 69.24 / 118
Misclassification error rate: 0.1527 = 20 / 131
a10 <-classError(p10, iter_t10,num) a10$misclassified 1 7 11 $errorRate 1 0.1538462 Classification tree:
tree(formula = class ~ ., data = iter7, na.action = na.pass,
split = c("gini"), x = FALSE, y = TRUE)
Variables actually used in tree construction:
1 "X219821_s_at" "X210387_at" "X221572_s_at" "X1564190_x_at"
5 "X228769_at" "X227094_at" "X227733_at"
Number of terminal nodes: 12
Residual mean deviance: 0.4563 = 54.3 / 119
Misclassification error rate: 0.1145 = 15 / 131
a7 <-classError(p7, iter_t7,num) a7$misclassified 1 3 10 $errorRate 1 0.1538462 Classification tree:
tree(formula = class ~ ., data = iter10, na.action = na.pass,
split = c("gini"), x = FALSE, y = TRUE)
Variables actually used in tree construction:
1 "X230134_s_at" "X228769_at" "X204712_at" "X227094_at" "X205695_at"
6 "X227356_at" "X1557483_at" "X223517_at"
Number of terminal nodes: 13
Residual mean deviance: 0.4802 = 56.66 / 118
Misclassification error rate: 0.1069 = 14 / 131
a10 <-classError(p10, iter_t10,num) a10$misclassified 1 7 9 $errorRate 1 0.1538462 Classification tree:
tree(formula = class ~ ., data = iter6, na.action = na.pass,
split = c("gini"), x = FALSE, y = TRUE)
Variables actually used in tree construction:
1 "X219821_s_at" "X219429_at" "X243476_at" "X228769_at" "X222283_at"
6 "X201611_s_at" "X1557483_at" "X227094_at"
Number of terminal nodes: 13
Residual mean deviance: 0.4657 = 54.96 / 118
Misclassification error rate: 0.1221 = 16 / 131
a6 <-classError(p6, iter_t6,num) a6$misclassified 1 5 10 $errorRate 1 0.1538462 Classification tree:
tree(formula = class ~ ., data = iter8, na.action = na.pass,
split = c("gini"), x = FALSE, y = TRUE)
Variables actually used in tree construction:
1 "X206766_at" "X219429_at" "X221572_s_at" "X226745_at" "X228769_at"
6 "X227356_at" "X236717_at" "X223147_s_at" "X226748_at" "X222657_s_at"
Number of terminal nodes: 13
Residual mean deviance: 0.473 = 55.82 / 118
Misclassification error rate: 0.1145 = 15 / 131
a8 <-classError(p8, iter_t8,num) a8$misclassified 1 5 11 $errorRate 1 0.1538462 |
Recent investments will yield a slight profit. |